一、公司情况
元构生物(Oristruct)是一家人工智能蛋白质设计开发应用企业,公司在相关技术创新领域储备深厚,并拥有来自中国科学技术大学且在蛋白质从头设计领域持续深耕多年的顶级技术团队。元构生物旨在通过人工智能提高蛋白质设计效率,构建“从头设计”平台,产出具有独立知识产权的全新或优化结构和功能的蛋白,未来用于“碳中和”、“生物医药”、“生物诊断”等场景。
本项目团队是国内外最早系统开展数据驱动蛋白质设计研究的团队之一。团队长期围绕蛋白质设计计算方法发展及实验验证开展研究,先后两次获中国科学技术大学年度杰出研究校长奖;发展的数据驱动蛋白质设计方法达到了国内外领先水平,构成了从头设计具有全新结构和序列的人工蛋白的完整工具链。该方法和工具获得广泛认可,被央视新闻联播、央视新闻、新华社、光明日报、人民网等多家媒体追踪报道,入选2022年度国内十大科技新闻等。
人工智能与蛋白质从头设计相结合,能够更高效的自动优化设计氨基酸序列和自动采样优化主链结构的统计能量模型和计算方法,对蛋白质的部分或全部进行从头设计,形成具有通用性的自动设计蛋白质结构和序列的工具链。并且能够用预先指定的特征作为约束条件高通量的生成待设计蛋白的主链空间结构,进而确定待设计蛋白的氨基酸序列,使待设计蛋白具有预先指定的特征,在绿色制造、酶设计、蛋白质药物设计等领域具有广泛的应用价值。
二、赋权项目技术及成果情况
刘海燕、陈泉团队长期深耕于数据驱动的蛋白质设计领域,前期工作中,建立并实验验证了一条全新的蛋白设计路线,包括利用神经网络能量函数从头设计主链结构的SCUBA模型(Nature 2022)、给定主链结构设计氨基酸序列的统计能量函数ABACUS(Bioinformatics 2020; Nature Communications 2014),及基于人工智能的序列设计方法ABACUS-R(Nature Computational Science 2022)。与现有参数化方法或基于天然蛋白质片段拼接方法不同,SCUBA方法原则上允许探索任意主链结构,并经过后续氨基酸序列设计获得能折叠成预期结构的人工蛋白。实验结果表明,SCUBA+ABACUS可设计新颖(自然界未被观察到)拓扑结构的全新蛋白质,提供了一条通用的蛋白质结构从头设计工具链。
具体如下:
1)建立了数据驱动的氨基酸序列设计方法ABACUS
如图1所示,对多个不同折叠类型的目标主链结构,用ABACUS设计的氨基酸序列能折叠成预期结构,且稳定性远超天然序列。至今为止,全自动设计蛋白质序列并实验验证成功的统计能量函数,国际上还未见报道,我们的工作表明统计能量模型在序列设计中具有重要价值。(Nature Communications 2014;Journal of Structural Biology 2016; Curr. Opin. Struct. Biol. 2016; Methods Mol. Biol. 2017)。改进后的ABACUS2在准确度和计算效率方面都有显著提高(Bioinformatics 2020)。结合ABACUS等算法可显著提高塑料降解酶PETase稳定性(ACS Catalysis, 2021)。
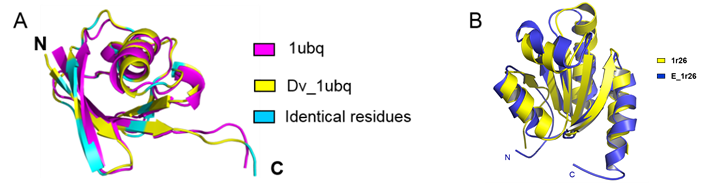
图1.基于ABACUS统计能量模型从头设计的序列Dv_1ubq(A)、E_1r26(B)与天然结构叠合图。
此外,我们建立了基于深度学习的氨基酸序列设计算法ABACUS-R,用Transformer 对每个中心残基所处的三维局部环境进行编码,并解码为中心残基类型,将该编码-解码器迭代应用于给定主链结构的多肽链上不同中心残基,得到自洽的整体序列。在实验验证中,ABACUS-R的设计成功率和设计精度超过了原有统计能量模型ABACUS,首次在实验结果上超越目前最优的序列从头设计算法(Nature Computational Science 2022)。
2)建立了数据驱动的主链结构从头设计方法SCUBA
在上述工作的基础上,我们基于统计能量函数建立了在序列待定条件下自动优化设计主链结构的SCUBA模型。理论计算和实验证明,用SCUBA设计主链结构,能够突破只能用天然片段来拼接产生新主链结构的限制,显著扩展从头设计蛋白的结构多样性,设计出不同于已知天然蛋白的新颖结构,是RosettaDesign之外目前唯一经充分实验验证的蛋白质从头设计方法,并与之互为补充。实验验证多个人工蛋白实际结构与设计模型一致,其中4个人工蛋白具有天然蛋白质中尚未观察到的新型拓扑结构(图2)。由于SCUBA能够在序列待定条件下连续搜索、优化主链结构,在功能蛋白设计更具有独特优势(Nature 2022)。
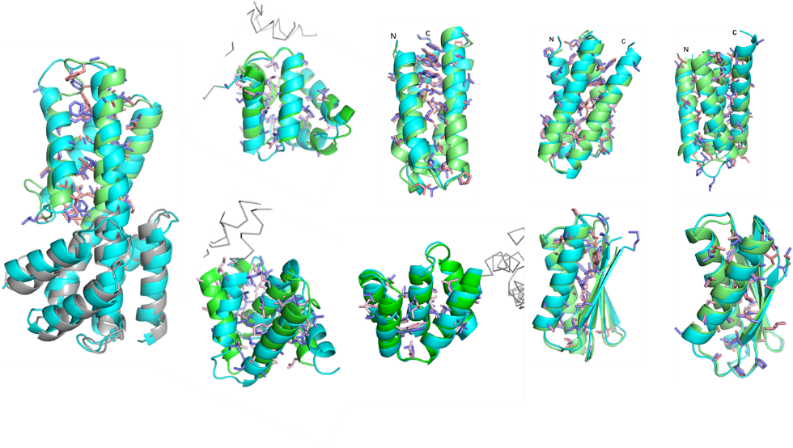
图2. 从头设计蛋白的高分辨晶体结构(天蓝色)与设计模型(绿色)比较。
3)建立了数据驱动的配体结合口袋设计方法
我们原创性建立了数据驱动的配体结合口袋设计方法DEPACT(Design Pocket as a Cluster based on Templates),并对DEPACT打分函数和设计口袋的合理性进行了评估。测试计算表明,无论目标小分子是否有已知的口袋结构信息,DEPACT方法均能针对其设计出合理的、特异性的口袋模型,并能够设计出近天然的水或金属离子介导的间接配位互作。在此基础上,结合侧链设计方法ABACUS2,采取匹配关键侧链原子的策略,我们建立了原创性口袋位置匹配方法PACMatch,此方法可兼顾侧链构象的合理性及与小分子互作的几何关系,实现复杂口袋和蛋白骨架之间的高效匹配,为快速、准确的酶分子活性位点匹配提供了高效方法 (J. Chem. Inf. Model. 2022)。以上数据驱动的主链骨架设计、序列设计、配体结合口袋设计、以及骨架匹配方法为新酶的从头设计奠定了理论基础。
4)建立了人工设计蛋白稳定性和相互作用的定向进化平台
我们首次提出将密西根大学Bardwell课题组开发的蛋白质稳定性筛选平台应用于从头设计蛋白的鉴定、筛选和改进,通过将蛋白质折叠稳定性与宿主的抗药性相关联,对理论设计序列可折叠性进行系统鉴定分析。对于设计不够理想的序列,可通过此平台进行定向进化优化,突变位点信息反馈设计,为改进设计方法提供可靠的实验反馈和指导。我们以Apaf-1 CARD结构域为目标从头设计的氨基酸序列设计结果不理想,经定向进化后获得多个抗性大幅提高的突变体,对其中两个突变体的三维结构进行了解析,其空间结构与设计目标高度一致(Nature Communications 2014)。将实验结果反馈设计,我们对设计方法ABACUS进行了改进。将优化后的设计方法应用于以硫氧还蛋白为模板的序列设计,获得了空间结构与设计模板高度一致的序列(Journal of Structural Biology 2016)。我们还基于此筛选平台对人工设计蛋白E_1r26进行了折叠性演化研究(Protein Science 2019)。
此外,我们成功建立了基于二氢叶酸还原酶DHFR拆分的蛋白质互补体系,用于鉴定和筛选蛋白-蛋白相互作用。在保留相互作用界面氨基酸的基础上,对其中一个蛋白其他残基进行序列重设计,获得了高热稳定性、且与设计模板亲和力相当的人工设计蛋白(Biotechnology and Bioengineering 2021)。此策略有望基于相互作用变化赋予蛋白质新的特性或调控功能,具有广阔的应用前景。
三、应用场景
数据驱动的AI蛋白质设计可以对蛋白质的部分或全部进行从头设计,形成具有通用性的自动设计蛋白质结构和序列,进而使待设计的蛋白具有预先指定的结构和功能特征。经过从头设计并验证功能的蛋白可以在碳中和、工业酶、蛋白质药物等多方面发挥巨大的应用价值。